写在前面
之前在投稿isbi时做对比实验的SSAE方法。近期有时间总结一下,防止后续需要的时候又找不到。
Stacked Sparse Autoencoder (SSAE) for Nuclei Detection on Breast Cancer Histopathology images
这篇isi热引论文采用堆叠稀疏自编码器(SSAE)对病理WSI做细胞核检测,虽然是检测,但也是通过滑动窗+图像块+细胞核/非细胞核二分类的思路实现。放在现在来看,方法比较简单,但是2013年投稿isbi2014的时候算是神经网络在计算病理领域的开创性工作。因此,决定好好理解一下AE,同时也matlab实现一下代码demo。
介绍
自编码器是神经网络的一种,经过无监督训练后可以将输入近似复制到输出。具体地,一个自编码器有两个部分:用于特征表示的编码器和用于生成重构的解码器,分别由函数$h = f(x)$和$r = g(h)$表示。其中,隐藏层$h$用来产生编码表示输入数据$^{[1]}$。具体的公式如下:
事实上,自编码器中,由输入到输出的复制并不太重要,重要的是我们通过这种方式,能够得到隐藏层$h$对于输入的特征编码。
极端来说,如果我们直接让$f()$和$g()$是线性函数,自编码器的输出值就会处处等于输入值,那这个特征编码就没有意义。因此,我们希望通过训练自编码器的输出值近似等于输入值,让隐藏层$h$能够学习到数据的有用特性来表征输入。而这通常需要向自编码器强加一些约束,使模型考虑输入的哪个部分需要被优先复制。
获取有用特征的一种方法是,限制隐藏层$h$的维度小于输入$x$的维度,即让数据有一定的损失后再进行重构恢复,使得自编码器学习数据中重要的特征。这种情况称为欠完备自编码器(undercomplete)。一般的,学习过程可以简单描述为最小化一个损失函数$L(x, g(f(x)))$。
通常地,我们可以直接通过对损失函数添加正则项来鼓励模型学习其他特性(如稀疏表示、对噪声的鲁棒性等),而不必一定需要隐藏层$h$的维度小于输入$x$的维度。实际中比较常见的有去噪自编码器和稀疏自编码器。
为了学习有用特征,去噪自编码器的做法是改变输入数据,引入噪声。它通过对原始数据$x$引入损坏过程$C(|)$得到$\tilde{x}$,再进行$g(f(\tilde{x}))$的变换,最后最小化损失函数$L(x, g(f(\tilde{x})))$的方式实现特征学习。
而稀疏自编码器的做法则是让隐藏层得到的特征更加稀疏 (也就是更多特征值接近0)。通过在损失函数中结合了重构误差$L(x,g(f(x)))$和稀疏正则项$\Omega(f(x))$的方式实现。
稀疏自编码器中的稀疏正则项
稀疏正则项是关于一个神经元的平均输出激活值的函数。对于第$i$个神经元,其平均输出激活定义为:
其中$n$是训练样本的数量,$x_j$是第$j$个训练样本,$w_{i}^{(1) T}$是权重矩阵$W^{(1)} $的第$i$行,$b_{i}^{(1)}$是$b^{(1)}$的第$i$个元素。
如果输出激活值很高,意味着神经元被“引爆”。而低的输出激活值意味着隐藏层神经元只对应小部分样本“引爆”。通过添加约束,使得$\hat{\rho}_{i}$较低,可以使自编码器学习一种每个神经元只“引爆”一小部分样本的特征表示。也就是说,每一个神经元通过响应一些只存在于一小部分训练样本中的特征来进行专门化。
而稀疏正则的目的,就是让输出激活值低,也就是约束隐藏层输出使其稀疏。具体做法为,在损失函数中加入正则项,当第$i$个神经元的平均激活值$\hat{\rho}_{i}$和希望的激活值${\rho}$差异很大时,给一个很大的惩罚值,反之,则较小的惩罚。一种惩罚方式是用KL散度度量。
用KL散度度量平均激活输出$\hat{\rho}_{i}$和期望输出${\rho}$的差异,当两者相同时,KLD为0。
这个时候,最小化损失函数不仅意味着最小化重构误差,也在最小化该正则项惩罚,使得$\hat{\rho}_{i}$和${\rho}$差异越来越小。其中,${\rho}$是训练稀疏自编码器时设置的超参数。
稀疏自编码器中的L2正则项
然而实际应用中,优化过程很可能出现$w^{(1)}$很大或者$z^{(1)}$很小,导致稀疏正则项很小。因此,通常地,我们会在损失函数中加一个L2权重正则项防止这种情况出现,定义为:
这里,$L$时隐藏层层数,$n$是样本数量,$k$是训练样本的维度。
稀疏自编码器的损失函数
此时,训练一个稀疏自编码器的损失函数是这样的:
其中,$\lambda$是L2正则项的系数,$\beta$是稀疏正则项的系数。在训练稀疏自编码器时,可以设置。
以下,我会讲解一个堆叠稀疏自编码器的实现demo。
代码详解1:载入数据及实例展示
载入matlab自带手写数字训练及测试数据集,其中image尺寸为28*28大小,以cell形式存储,label以one-hot格式存储。另外,这里随机选取训练数据中的9个实例进行可视化。
代码详解2:第一个AE训练及超参数设置
对于28*28大小的图像数据(直接拉成784维特征),先采用一个稀疏自编码器进行训练用来特征提取。
其中,参数设置:隐藏层特征200维,迭代训练500次,L2权重正则化系数0.004,稀疏正则系数4,稀疏值0.15。
matlab的toolbox做得很好,训练过程可视化很清晰。包括自编码器网络可视化、进程以及实时performance。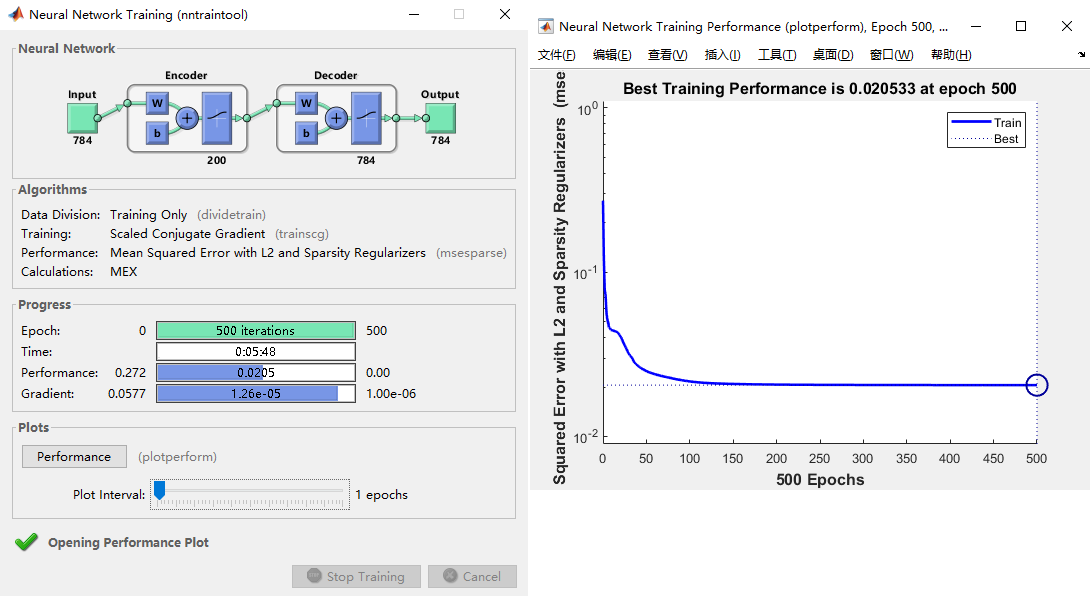
训练完成后,得到自编码器模型$autoenc1$。
代码详解3:模型参数可视化和重构结果展示
|
对于训练好的模型,我们可以采用plotWeights对模型中的编码器进行权重可视化。
同样,为了直观查看自编码模型的好坏,我们可以将该模型应用于测试集predict得到所有图像对应的重构输出,然后show出来进行定性评估。
代码详解4:特征提取
尽管直观来看,我们经过一次的重构结果已经不错,但是对于SSAE来说,我们希望有多个堆叠的自编码器进行特征提取。也就是说,第一个自编码器模型的解码器部分只是用来训练重构误差,而编码器部分才是用来对输入进行特征提取的关键。我们将第一个自编码器的编码部分encode拿出来对训练集数据进行特征提取,然后将提取的特征用于后续第二个自编码器的输入。
代码详解5:第二个AE训练及超参数设置
对于$autoenc1$提取到的200维的特征$feat1$,采用第二个稀疏自编码器进行训练用来进一步地特征提取。
其中,参数设置:隐藏层特征100维,迭代训练500次,L2权重正则化系数0.002,稀疏正则系数0.4,稀疏值0.1。
AE训练过程可视化如图所示: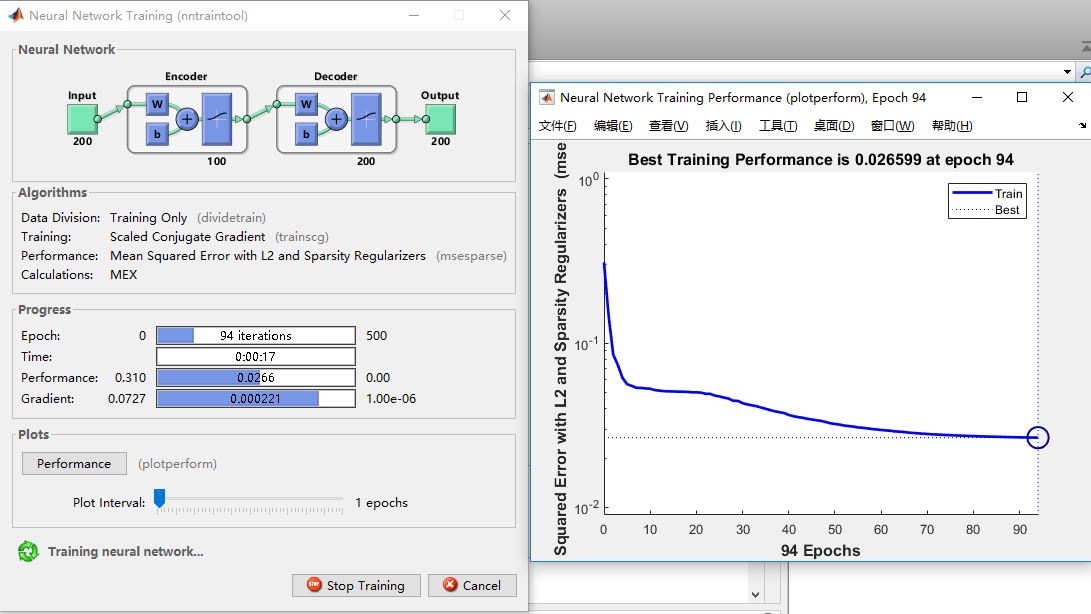
代码详解6:特征提取及分类器训练
训练完成后,得到第二个自编码器模型$autoenc2$。此时我们将第二个自编码器的编码部分encode拿出来再次对$feat1$进行特征提取得到$feat2$。此时的$feat2$即代表了我们用堆叠自编码器提取到的图像的特征。也就是说,每一张28*28大小的图像被100维特征表示。
此时我们可以用trainsoftmaxLayer分类器进行有监督地训练。得到分类器模型$softmax$。
softmax训练过程可视化如图所示: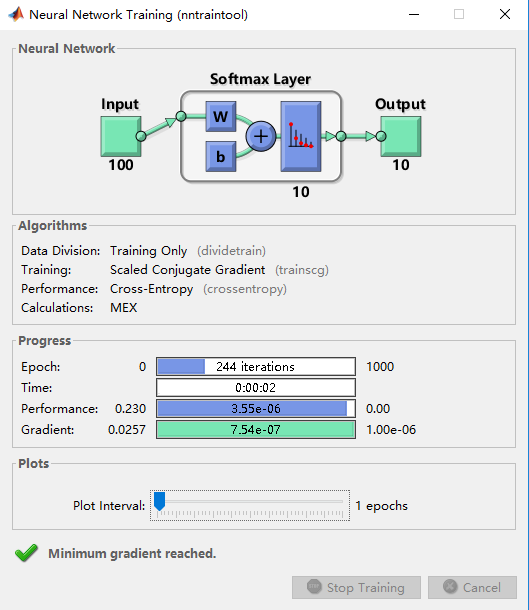
|
代码详解7:样本测试及定量评估
得到分类器模型,我们可以进行测试样本的评估。先对测试图像应用$autoenc1$提取200维特征,然后$autoenc2$对这200维特征提取100维特征$testfeats2$并送入$softmax$分类器得到样本的预测结果$pred$(以softmax概率形式)。
可以用matlab内置的plotconfusion函数直接得到混淆矩阵,如图。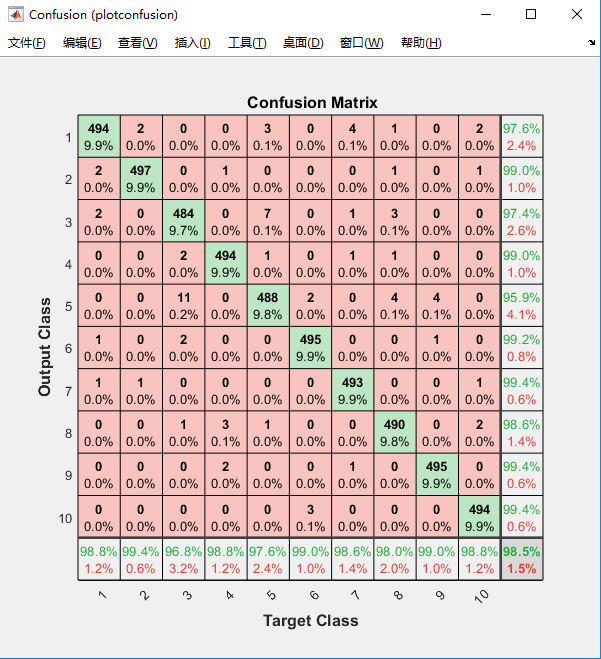
|
代码详解8:可视化整个网络结构
整个SSAE的过程大致如此。为了直观理解我们整个过程用了哪些网络结构,我们还可以将所有的模型结构stack起来view: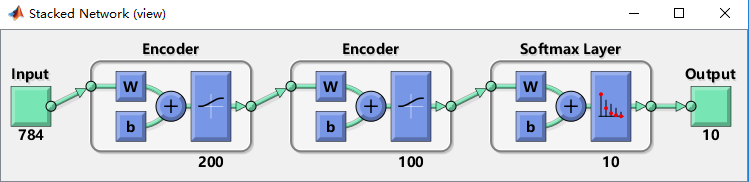
代码总结
SSAE的代码就是这样,比较简单,而且matlab的toolbox集成相当好,整个过程很直观。当然,该demo也还有很多可以自行开发的地方:
- 数据:这里只是展示SSAE应用到matlab自带手写数字集上的demo,完全可以迁移到自己的数据集上进行分类应用。具体的数据制作方法,可以见下面的附录function:数据生成;
- 特征提取: 稀疏自编码器(SAE)的本质是无监督提取特征,因此可以更换其他特征提取方法或者其他自编码器();
- 分类器:softmax分类器其实是一种神经网络方法。这里的分类器和SSAE部分是相互独立的。可以换成其他分类器,如SVM,LDA等等。乃至换成无监督聚类也未尝不可。
附录function: 数据生成
|
参考
[1] 深度学习 自编码器章节Ian Goodfellow
[2] 自编码器是什么?
[3] 为什么稀疏自编码器很少见到多层的?
[4] Matlab中的trainAutoencoder